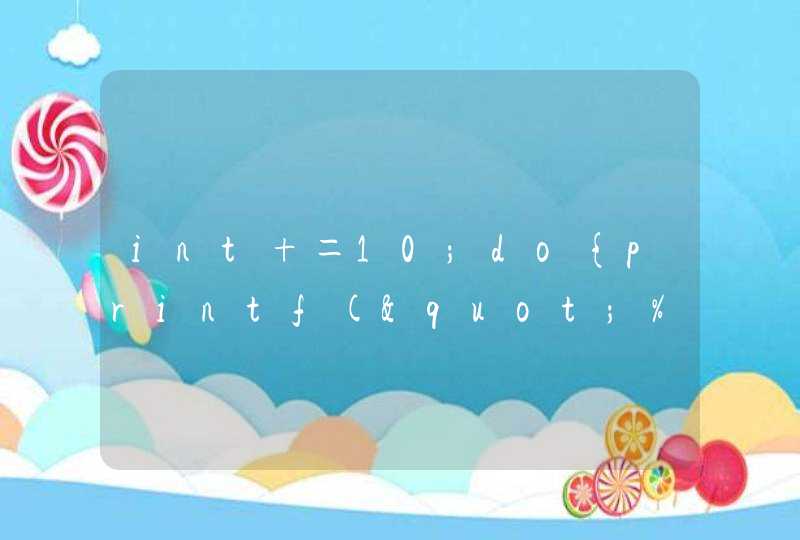如data.frame为:zz, 绘图如下:
a. single protein:线性回归画法
1. ggplot(zz,aes(x=a, y=HDL))+
geom_point(alpha=1,colour="#FFA54F")+
geom_smooth(method = lm,colour="#8B658B")+
#scale_color_brewer(palette = "Set1")+
theme_bw()+
labs(x="Ferritin",y="HDL.C",title="Pearson’s correlation test of ferritin and HDL.C")+
annotate("text", x = 1000, y = 2.5, label = "r = -0.51",colour="black",size=4)
2. library(ggstatsplot)
ggscatterstats(data = alldata,
y = TRANSFUSION.UNIT,
x = NPTXR,
centrality.para = "mean", #"mean" or "median"
margins = "both",
xfill = "#D8BFD8",
yfill = "#EEDD82",
#line.size= ,
line.color="#8B6969",
point.color="#2F4F4F",
marginal.size=4,
marginal.type = "density", # "histogram", "boxplot", "density", "violin", "densigram")
title = "Relationship between TRANSFUSION.UNIT and NPTXR")
b. ggcorrplot, 全部蛋白 global correlation map 画法
ggcorrplot(cor(alldata))
2. summary(lm(y~x),method=" ") %>%.[["coefficients"]] 正规线性回归
(其实就是:a<-lm(y~x1+x2+...,data)
plot(summary(lm(y~x),method=" ")) #绘图
3. ggcor部分数据绘图: 数据类型为data.frame,纵坐标为各指标or各蛋白,行为观测值。
data <- fortify_cor(alldata[,10:11],alldata,cluster.type = "col")
ggcor<-ggcor(data,label_size=0.5) +
geom_colour()+
theme(axis.text.x = element_text(colour = "black",size = 4.7),
axis.text.y=element_text(size=5.5),
axis.ticks=element_blank())+
geom_num(aes(num=r),colour="black",size=1.5)
4. corrr包画法
datasets::mtcars %>%
correlate() %>%
focus(-cyl, -vs, mirror = TRUE) %>%
rearrange() %>%
network_plot(min_cor = .2)
相关性分析用于评估两个或多个变量之间的关联性。
皮尔森相关性分析是一种参数相关性检验,检测的是两个变量间的线性关系;应用皮尔森相关性分析的前提是两个变量都是正态分布的,其相关性可以用线性回归曲线表示。
H0: true correlation is equal to 0
cor.test(){stats} 返回一系列参数,主要关注p.value 和 correlation coefficient(ample estimates: cor );
可以改变cor.test(){stats}中的method 参数进行非参检验,但stats 的作者都表示用上面提到的包更的支持数据种类更多、估计结果准确性更高。
实际上就是每一个变量与其他变量间的相关性检验,因此方法也是上面提到的参数相关和非参相关检验。
多个检验同时进行时,如果对任意单个假设检验问题,p-值小于 α就拒绝原假设,则无法控制总体第一类错误率(family-wise error rate, FWER);FWER随检验个数 mm增大而增大( m→∞m→∞时收敛到1)。
总的来说,当同一个数据集有n次(n>=2)假设检验时,就要做多重假设检验校正
p.adjust(){stats}中的参数: p.adjust.methods = c("holm", "hochberg", "hommel", "bonferroni", "BH", "BY","fdr", "none")
Hochberg's and Hommel's : 适用于假设检验是独立或非负相关的检验结果, Hommel的方法比Hochberg的方法更强大,但差异通常很小,而Hochberg 的计算速度更快。
* BH{aka fdr(False Discovery Rate)} : 是控制错误发现率,即将假阳性结果和真阳性的比例控制在一定范围内。 错误发现率是一种不如第一类错误率(family-wise error rate, FWER)严格的条件,因此这些方法比其他方法更有效,也是非常常用的方法。
bonferroni :通过对p值的阈值进行校正来实现消除假阳性结果,是最严格的矫正方法,校正后拒绝的不只是假阳性结果,很多阳性结果也会被它拒绝。bonferroni 通过公式 p*(1/n){其中p为原始阈值,n为总检验次数},拒绝le所有的假阳性结果发生的可能性。
H0: 两矩阵没有相关关系。
在微生物群落分析中, 免不了分析环境因子与菌落的相关性,此时便需要做CCA 和 Mantel test 分析 。Mantel test 分析是将微生物群落作为一个距离矩阵(如UniFrac distance matrix),环境变量作为另一个距离矩阵(如pH、有机碳、总氮、盐度、温度、地理等),再检验两个矩阵之间的相关性。
mantel.rtest (){ ade4 }
mantel(){ecodist}
mantel(){vegan}
ggcor不仅内置了mental test 函数, 也很好的实现了mental test 可视化。
如果是矩阵相关系数计算,结果可能不怎么好解读。通过热图的方式可以有效的将结果分为多个层次,然后再对每个层次进行专注解读就显得方便多了。
专门为相关矩阵可视化写的R包也不少,包括画风比较粗狂、但结果又比较详细的corrgram, GGally, PerformanceAnalytics等,他们可以将原始数据分布,相关系数,线性回归的回归线,显著性P值等展示在一张画布中;而基于base绘图系统写的 corrplot 应该是最为精美的了,配色清新,功能齐全。对于已经习惯用了grid 图形系统的ggplot2语法的人来说, ggcorrplot 只实现了小部分的corrplot内容,虽然也很精美但是又有些意犹未尽的感觉;还在紧锣密鼓构建中的 ggcor 将满足大部分的相关系数可视化需求。
ggcor不同于常规的ggplot2扩展包,它引入了ggcor函数,目的调用ggplot() 来进行图层初始化,因此很多图层参数是通用的;但它需要相关系数矩阵来进行数据处理、绘图类型、背景、坐标轴、颜色映射、图例设置等,因此矩阵需要在这一步就输入。要么调用作者封装的cor(){stats}或 cor.test(){stats},要么处理数据后用 as_cor_tbl() 和 fortify_cor()两个函数导入,应该能满足基本需求。
as_cor_tbl()函数 :
fortify_cor()函数 :
主要用于处理原始数据表,可以调用cor(){stats}求相关系数,默认使用pearson方法,当然spearman和kendall方法也都支持。但stats 包的作者在cor() 下面提到,如果要用spearman和kendall 的方法,最好用其他的包。而且涉及P值矫正什么的,可能cor() 或者 cor.test() 函数并不能达到要求,最好还是自己做统计分析,最后进行数据格式处理。
几个重要图层:
geom_square()、geom_circle2()、geom_ellipse2()、geom_pie2()、geom_colour()、geom_confbox()、geom_num()、geom_mark()、geom_cross()
基本就是形状、色彩、大小等,值得提出来说的只有geom_cross() 这个是根据阈值,在阈值外的位置打上一个X。