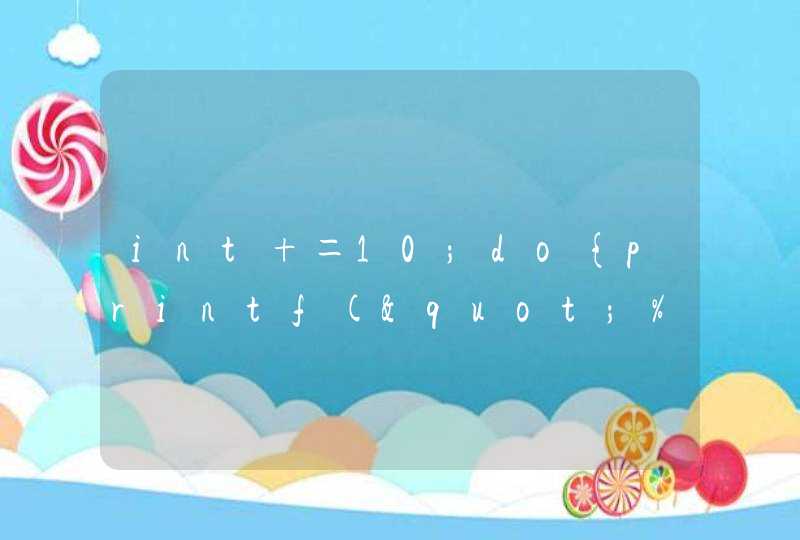排课问题的本质是将课程、教师和学生在合适的时间段内分配到合适的教室中,涉及到的因素较多,是一个多目标的调度问题,在运筹学中被称为时间表问题(Timetable Problem,TTP)。设一个星期有n个时段可排课,有m位教师需要参与排课,平均每位教师一个星期上k节课,在不考虑其他限制的情况下,能够推出的可能组合就有 种,如此高的复杂度是目前计算机所无法承受的。因此众多研究者提出了多种其他排课算法,如模拟退火,列表寻优搜索和约束满意等。
Github : https://github.com/xiaochus/GeneticClassSchedule
遗传算法(Genetic Algorithm)是模拟达尔文生物进化论的自然选择和遗传学机理的生物进化过程的计算模型,是一种通过模拟自然进化过程搜索最优解的方法。遗传算法的流程如下所示:
遗传算法首先针对待解决问题随机生成一组解,我们称之为种群(Population)。种群中的每个个体都是问题的解,在优化的过程中,算法会计算整个种群的成本函数,从而得到一个与种群相关的适应度的序列。如下图所示:
为了得到新的下一代种群,首先根据适应度对种群进行排序,从中挑选出最优的几个个体加入下一代种群,这一个过程也被称为精英选拔。新种群余下的部分通过对选拔出来的精英个体进行修改得到。
对种群进行修改的方法参考了生物DAN进化的方法,一般使用两种方法: 变异 和 交叉 。 变异 的做法是对种群做一个微小的、随机的改变。如果解的编码方式是二进制,那么就随机选取一个位置进行0和1的互相突变;如果解的编码方式是十进制,那么就随机选取一个位置进行随机加减。 交叉 的做法是随机从最优种群中选取两个个体,以某个位置为交叉点合成一个新的个体。
经过突变和交叉后我们得到新的种群(大小与上一代种群一致),对新种群重复重复上述过程,直到达到迭代次数(失败)或者解的适应性达到我们的要求(成功),GA算法就结束了。
算法实现
首先定义一个课程类,这个类包含了课程、班级、教师、教室、星期、时间几个属性,其中前三个是我们自定义的,后面三个是需要算法来优化的。
接下来定义cost函数,这个函数用来计算课表种群的冲突。当被测试课表冲突为0的时候,这个课表就是个符合规定的课表。冲突检测遵循下面几条规则:
使用遗传算法进行优化的过程如下,与上一节的流程图过程相同。
init_population :随机初始化不同的种群。
mutate :变异操作,随机对 Schedule 对象中的某个可改变属性在允许范围内进行随机加减。
crossover :交叉操作,随机对两个对象交换不同位置的属性。
evolution :启动GA算法进行优化。
实验结果
下面定义了3个班,6种课程、教师和3个教室来对排课效果进行测试。
优化结果如下,迭代到第68次时,课程安排不存在任何冲突。
选择1203班的课表进行可视化,如下所示,算法合理的安排了对应的课程。
遗传算法(GA)是最早由美国Holland教授提出的一种基于自然界的“适者生存,优胜劣汰”基本法则的智能搜索算法。遗传算法也是借鉴该基本法则,通过基于种群的思想,将问题的解通过编码的方式转化为种群中的个体,并让这些个体不断地通过选择、交叉和变异算子模拟生物的进化过程,然后利用“优胜劣汰”法则选择种群中适应性较强的个体构成子种群,然后让子种群重复类似的进化过程,直到找到问题的最优解或者到达一定的进化(运算)时间。
首先遗传算法是一种优化算法,通过模拟基因的优胜劣汰,进行计算(具体的算法思路什么的就不赘述了)。大致过程分为初始化编码、个体评价、选择,交叉,变异。
以目标式子 y = 10 * sin(5x) + 7 * cos(4x)为例,计算其最大值
首先是初始化,包括具体要计算的式子、种群数量、染色体长度、交配概率、变异概率等。并且要对基因序列进行初始化
[python] view plain copy
pop_size = 500 # 种群数量
max_value = 10 # 基因中允许出现的最大值
chrom_length = 10 # 染色体长度
pc = 0.6 # 交配概率
pm = 0.01 # 变异概率
results = [[]] # 存储每一代的最优解,N个二元组
fit_value = [] # 个体适应度
fit_mean = [] # 平均适应度
pop = geneEncoding(pop_size, chrom_length)
其中genEncodeing是自定义的一个简单随机生成序列的函数,具体实现如下
[python] view plain copy
def geneEncoding(pop_size, chrom_length):
pop = [[]]
for i in range(pop_size):
temp = []
for j in range(chrom_length):
temp.append(random.randint(0, 1))
pop.append(temp)
return pop[1:]
编码完成之后就是要进行个体评价,个体评价主要是计算各个编码出来的list的值以及对应带入目标式子的值。其实编码出来的就是一堆2进制list。这些2进制list每个都代表了一个数。其值的计算方式为转换为10进制,然后除以2的序列长度次方减一,也就是全一list的十进制减一。根据这个规则就能计算出所有list的值和带入要计算式子中的值,代码如下
[python] view plain copy
# 0.0 coding:utf-8 0.0
# 解码并计算值
import math
def decodechrom(pop, chrom_length):
temp = []
for i in range(len(pop)):
t = 0
for j in range(chrom_length):
t += pop[i][j] * (math.pow(2, j))
temp.append(t)
return temp
def calobjValue(pop, chrom_length, max_value):
temp1 = []
obj_value = []
temp1 = decodechrom(pop, chrom_length)
for i in range(len(temp1)):
x = temp1[i] * max_value / (math.pow(2, chrom_length) - 1)
obj_value.append(10 * math.sin(5 * x) + 7 * math.cos(4 * x))
return obj_value
有了具体的值和对应的基因序列,然后进行一次淘汰,目的是淘汰掉一些不可能的坏值。这里由于是计算最大值,于是就淘汰负值就好了
[python] view plain copy
# 0.0 coding:utf-8 0.0
# 淘汰(去除负值)
def calfitValue(obj_value):
fit_value = []
c_min = 0
for i in range(len(obj_value)):
if(obj_value[i] + c_min > 0):
temp = c_min + obj_value[i]
else:
temp = 0.0
fit_value.append(temp)
return fit_value
然后就是进行选择,这是整个遗传算法最核心的部分。选择实际上模拟生物遗传进化的优胜劣汰,让优秀的个体尽可能存活,让差的个体尽可能的淘汰。个体的好坏是取决于个体适应度。个体适应度越高,越容易被留下,个体适应度越低越容易被淘汰。具体的代码如下
[python] view plain copy
# 0.0 coding:utf-8 0.0
# 选择
import random
def sum(fit_value):
total = 0
for i in range(len(fit_value)):
total += fit_value[i]
return total
def cumsum(fit_value):
for i in range(len(fit_value)-2, -1, -1):
t = 0
j = 0
while(j <= i):
t += fit_value[j]
j += 1
fit_value[i] = t
fit_value[len(fit_value)-1] = 1
def selection(pop, fit_value):
newfit_value = []
# 适应度总和
total_fit = sum(fit_value)
for i in range(len(fit_value)):
newfit_value.append(fit_value[i] / total_fit)
# 计算累计概率
cumsum(newfit_value)
ms = []
pop_len = len(pop)
for i in range(pop_len):
ms.append(random.random())
ms.sort()
fitin = 0
newin = 0
newpop = pop
# 转轮盘选择法
while newin < pop_len:
if(ms[newin] < newfit_value[fitin]):
newpop[newin] = pop[fitin]
newin = newin + 1
else:
fitin = fitin + 1
pop = newpop
以上代码主要进行了3个操作,首先是计算个体适应度总和,然后在计算各自的累积适应度。这两步都好理解,主要是第三步,转轮盘选择法。这一步首先是生成基因总数个0-1的小数,然后分别和各个基因的累积个体适应度进行比较。如果累积个体适应度大于随机数则进行保留,否则就淘汰。这一块的核心思想在于:一个基因的个体适应度越高,他所占据的累计适应度空隙就越大,也就是说他越容易被保留下来。选择完后就是进行交配和变异,这个两个步骤很好理解。就是对基因序列进行改变,只不过改变的方式不一样
交配:
[python] view plain copy
# 0.0 coding:utf-8 0.0
# 交配
import random
def crossover(pop, pc):
pop_len = len(pop)
for i in range(pop_len - 1):
if(random.random() < pc):
cpoint = random.randint(0,len(pop[0]))
temp1 = []
temp2 = []
temp1.extend(pop[i][0:cpoint])
temp1.extend(pop[i+1][cpoint:len(pop[i])])
temp2.extend(pop[i+1][0:cpoint])
temp2.extend(pop[i][cpoint:len(pop[i])])
pop[i] = temp1
pop[i+1] = temp2
变异:
[python] view plain copy
# 0.0 coding:utf-8 0.0
# 基因突变
import random
def mutation(pop, pm):
px = len(pop)
py = len(pop[0])
for i in range(px):
if(random.random() < pm):
mpoint = random.randint(0, py-1)
if(pop[i][mpoint] == 1):
pop[i][mpoint] = 0
else:
pop[i][mpoint] = 1
整个遗传算法的实现完成了,总的调用入口代码如下
[python] view plain copy
# 0.0 coding:utf-8 0.0
import matplotlib.pyplot as plt
import math
from calobjValue import calobjValue
from calfitValue import calfitValue
from selection import selection
from crossover import crossover
from mutation import mutation
from best import best
from geneEncoding import geneEncoding
print 'y = 10 * math.sin(5 * x) + 7 * math.cos(4 * x)'
# 计算2进制序列代表的数值
def b2d(b, max_value, chrom_length):
t = 0
for j in range(len(b)):
t += b[j] * (math.pow(2, j))
t = t * max_value / (math.pow(2, chrom_length) - 1)
return t
pop_size = 500 # 种群数量
max_value = 10 # 基因中允许出现的最大值
chrom_length = 10 # 染色体长度
pc = 0.6 # 交配概率
pm = 0.01 # 变异概率
results = [[]] # 存储每一代的最优解,N个二元组
fit_value = [] # 个体适应度
fit_mean = [] # 平均适应度
# pop = [[0, 1, 0, 1, 0, 1, 0, 1, 0, 1] for i in range(pop_size)]
pop = geneEncoding(pop_size, chrom_length)
for i in range(pop_size):
obj_value = calobjValue(pop, chrom_length, max_value) # 个体评价
fit_value = calfitValue(obj_value) # 淘汰
best_individual, best_fit = best(pop, fit_value) # 第一个存储最优的解, 第二个存储最优基因
results.append([best_fit, b2d(best_individual, max_value, chrom_length)])
selection(pop, fit_value) # 新种群复制
crossover(pop, pc) # 交配
mutation(pop, pm) # 变异
results = results[1:]
results.sort()
X = []
Y = []
for i in range(500):
X.append(i)
t = results[i][0]
Y.append(t)
plt.plot(X, Y)
plt.show()
最后调用了一下matplotlib包,把500代最优解的变化趋势表现出来。完整代码可以在github 查看
欢迎访问我的个人博客
阅读全文