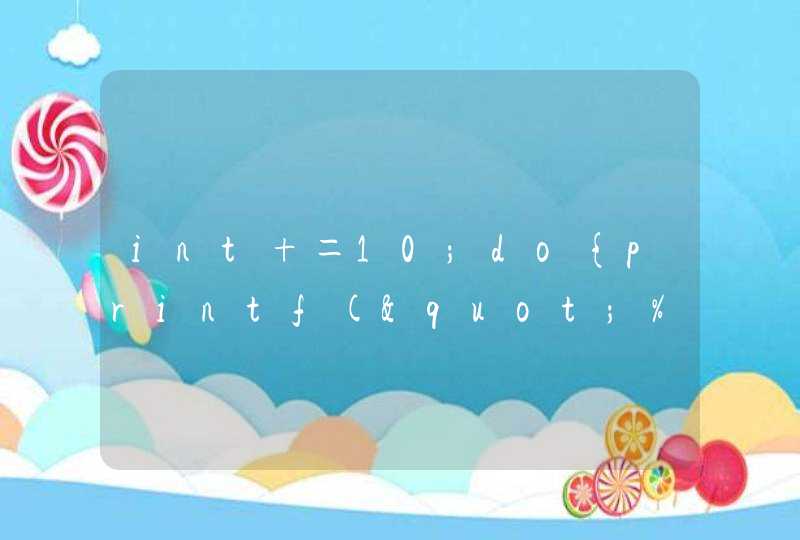d1 <- do.call(cbind, lapply(lapply(d, unlist), length<- , max(lengths(d))))
lapply(x,fun),lapply接收两个值,x为向量或列表,fun是方法,这个函数的意思是将fun方法作用到x的每个元素,返回一个与x相同长度的列表
unlist函数接收一个列表,unlist将其简化为一个包含列表中所有原子成分的向量
外面嵌套的那层lapply函数,是将每个向量元素的长度设定为最长的那个长度,缺失值用NA补齐
cbind是按列合并向量,如果两列数量不一样会自动重复短的那列
do.call函数与lapply函数功能类似,但有一点不一样
lapply()为列表中的每个元素应用一个给定的函数,所以会有几个函数调用。
do.call()将给定的函数作为一个整体应用于列表,所以只有一个函数调用。
详细差别见 http://www.dovov.com/rlapplydo-call.html
a本身是一个矩阵,而定义dimnames=list()则表示其每一个元素都被命名且命名方式是列表(list),因此在调用a中的元素的时候可以调用a[]或者a[[]]都可以。a[]是调用a本身的第几个元素,a[[]]是命名中的第几个名字下的元素。请问大家,在运行R语言的时候,出现了以下问题,不知道是源文件的问题,还是程序的问题,求解
源文件如下
There were 50 or more warnings (use warnings() to see the first 50)
#install.packages("survival")
>
>library(survival) #引用包
>inputFile="expTime.txt" #输入文件
>pFilter=0.001 #显著性过滤条件
>setwd("C:\\Users\\ASUS\\Desktop\\tmeimmune\\22.cox") #设置共工作目录
>
>#读取输入文件
>rt=read.table(inputFile,header=T,sep="\t",check.names=F,row.names=1)
>rt$futime=rt$futime/365 #以年为单位,除以365
>
>outTab=data.frame()
>for(gene in colnames(rt[,3:ncol(rt)])){
+ #单因素cox分析
+ cox=coxph(Surv(futime, fustat) ~ rt[,gene], data = rt)
+ coxSummary = summary(cox)
+ coxP=coxSummary$coefficients[,"Pr(>|z|)"]
+
+ #KM检验,后续需要可视乎,要和生存相关
+ group=ifelse(rt[,gene]<=median(rt[,gene]),"Low","High")
+ diff=survdiff(Surv(futime, fustat) ~ group,data = rt)
+ pValue=1-pchisq(diff$chisq,df=1)
+
+ #保存满足条件的基因
+ if((pValue<pFilter) &(coxP<pFilter)){
+ outTab=rbind(outTab,
+ cbind(gene=gene,
+ KM=pValue,
+ HR=coxSummary$conf.int[,"exp(coef)"],
+ HR.95L=coxSummary$conf.int[,"lower .95"],
+ HR.95H=coxSummary$conf.int[,"upper .95"],
+ pvalue=coxP) )
+ }
+ }
There were 50 or more warnings (use warnings() to see the first 50)
>#输出基因和P值表格文件
>write.table(outTab,file="cox.result.txt",sep="\t",row.names=F,quote=F)
>
>#绘制森林图
>#读取输入文件
>rt <- read.table("cox.result.txt",header=T,sep="\t",row.names=1,check.names=F)
Error in read.table("cox.result.txt", header = T, sep = "\t", row.names = 1, :
no lines available in input
>gene <- rownames(rt)
>hr <- sprintf("%.3f",rt$"HR")
>hrLow <- sprintf("%.3f",rt$"HR.95L")
>hrHigh <- sprintf("%.3f",rt$"HR.95H")
>Hazard.ratio <- paste0(hr,"(",hrLow,"-",hrHigh,")")
>pVal <- ifelse(rt$pvalue<0.001, "<0.001", sprintf("%.3f", rt$pvalue))
>
>#输出图形
>pdf(file="forest.pdf", width = 7,height = 6)
>n <- nrow(rt)
>nRow <- n+1
>ylim <- c(1,nRow)
>layout(matrix(c(1,2),nc=2),width=c(3,2))
>
>#绘制森林图左边的基因信息
>xlim = c(0,3)
>par(mar=c(4,2.5,2,1))
>plot(1,xlim=xlim,ylim=ylim,type="n",axes=F,xlab="",ylab="")
>text.cex=0.8
>text(0,n:1,gene,adj=0,cex=text.cex)
>text(1.5-0.5*0.2,n:1,pVal,adj=1,cex=text.cex)text(1.5-0.5*0.2,n+1,'pvalue',cex=text.cex,font=2,adj=1)
Error in text.default(1.5 - 0.5 * 0.2, n:1, pVal, adj = 1, cex = text.cex) :
'labels'长度不能设成零
>text(3,n:1,Hazard.ratio,adj=1,cex=text.cex)text(3,n+1,'Hazard ratio',cex=text.cex,font=2,adj=1,)
>
>#绘制森林图
>par(mar=c(4,1,2,1),mgp=c(2,0.5,0))
>xlim = c(0,max(as.numeric(hrLow),as.numeric(hrHigh)))
Warning message:
In max(as.numeric(hrLow), as.numeric(hrHigh)) :
no non-missing arguments to maxreturning -Inf
>plot(1,xlim=xlim,ylim=ylim,type="n",axes=F,ylab="",xaxs="i",xlab="Hazard ratio")
Error in plot.window(...) : 'xlim'值不能是无限的
>arrows(as.numeric(hrLow),n:1,as.numeric(hrHigh),n:1,angle=90,code=3,length=0.05,col="darkblue",lwd=2.5)
Error in arrows(as.numeric(hrLow), n:1, as.numeric(hrHigh), n:1, angle = 90, :
不能将零长度的座标同其它长度的座标混合在一起
>abline(v=1,col="black",lty=2,lwd=2)
>boxcolor = ifelse(as.numeric(hr) >1, 'red', 'green')
>points(as.numeric(hr), n:1, pch = 15, col = boxcolor, cex=1.3)
Error in xy.coords(x, y) : 'x' and 'y' lengths differ
>axis(1)
>dev.off()
null device
1