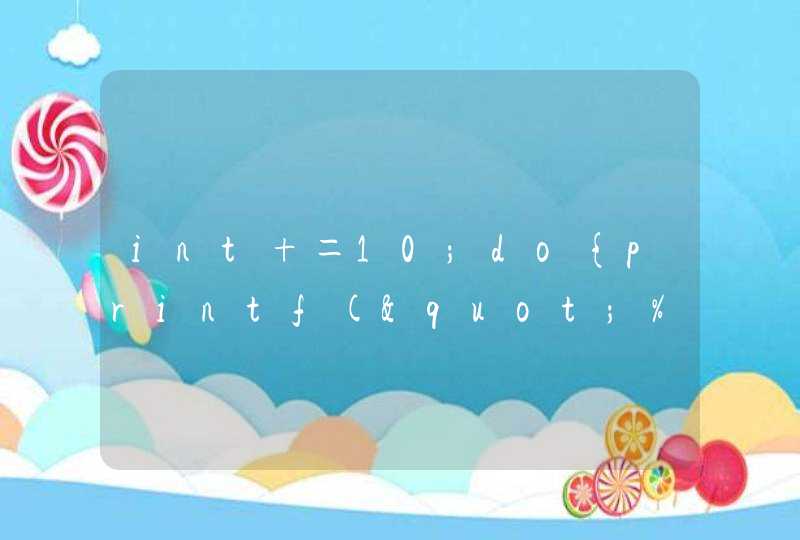想获取R语言相关系数meta分析的程序模板的同学请在公众号(全哥的学习生涯)内回复“相关系数”即可。
meta数据包提供实现相关系数的Meta分析命令是:metacor(),这个命令通过加权的倒方差法运用相关系数和纳入的样本数来实现相关系数的随机效用模型和固定效用模型的合并,得到合并的相关系数及95%可信区间。具体的命令如下:
metacor(cor, n,studlab, data= NULL, subset=NULL, sm=.settings$smcor)
cor为每一个纳入研究的相关系数, n为样本量, studlab纳入研究的标签向量, data为相应的的数据集,sm选项为合并的方法,包括ZCOR和COR,其中ZCOR是合并之前先做Fisher Z变换,COR是直接合并。具体的步骤如下:
library(meta)
data<-read.csv(“C:/Users/86187/Desktop/data.csv”)
录入的数据见图1。
data<-metacor(r,n,data=m1,sm="ZCOR")
在这里合并的方法用的是Fisher Z变换。对样本的相关系数做Fisher Z变换是因为Fisher Z变换可以使样本的相关系数的分布正态分布,尤其是在样本量较小的时候,这样便于进一步估计。一般来说,不管是随机还是固定效应都会先对相关系数做Fisher Z变换。只有很少的情况下才直接用相关系数直接来做分析,比如样本量很大的时候,如果直接合并相关系数,当相关系数值接近1的时候,小样本量研究得到的权重会非常大。因此在这里推荐合并的方法都用(ZCOR)Fisher Z变换。Meta分析的结果见图2。
结果显示,异质性检验Q=6.16, P=0.0461, I2=67.5,可以认为有统计学意义上的异质性。选用随机效用模型,COR=0.8427, 95%CI: 0.6264-0.9385, z=4.8724, P<0.0001, 有统计学差异。
具体的命令如下:
forest(a)
从森林图中,非常简单和直观地看到Meta分析的统计结果,见图3
关于这两个方法的介绍请看我之前公众号(全哥的学习生涯)的推送文章(如何用R语言进行meta分析,详细教程一)的内容。敏感性分析和剪补法的结果图分别见图4和图5。
通常Meta分析假定效应量来自于独立的研究,因此统计结果也是独立的。然而,许多研究不能满足独立性的假设,比如多个治疗组与一个共同的对照组比较的研究和多个结局变量的研究就可能产生效应量之间的相关。多变量meta 分析(multivariate meta⁃analysis)作为单变量meta分析的一个拓展,可合并估计多个研究的多个相关参数,这些参数可以是多个结局或多组间的比较。当同一总体中的测量结局相关时,分别对每个结局进行Meta 分析,测量结局之间的相关结构就可能被忽略。多变量Meta分析在随机对照研究中有多种应用,最简单的是在临床试验中把每个组的结局分别处理,其他的应用还有同时探索两个临床结局的治疗效应,或同时探索成本效益的治疗效应,比较多个治疗的联合试验,以及在观察性研究中评估暴露量与疾病之间的相关性,还有在诊断试验和网络干预中的应用。
本次数据来源请见文末的参考文献,主要研究肝硬化的非手术治疗方式预防其出血的危险性,以初次出血的例数为指标,其中三个组分别是:β⁃受体阻滞剂(A),硬化疗法(B),对照组(C),目的是评价这三种非手术治疗方式预防肝硬化出血的效果。,Bled表示初次出血的例数,Total表示干预组的总例数。YAC和YBC分别表示A、B两组相对于C组估计的ln(OR),即干预组的肝硬化初次出血的危险性是对照组的倍数的自然对数;SAA、SBB和SAB则表示其对应方差及两者之间的协方差。对于包含0的研究(研究10和研究20),在每个组增加0.5个初次出血的例数。整理后见表1。
随后安装调用程序包,并进行加载:
install.packages(‘mvmeta’)
library(mvmeta)。
随后将肝硬化初次出血整理后的数据集data(至少包含YAC、YBC、SAA、SAB、SBB变量)保存为csv格式,然后利用下面命令将其导入R语言。
mvmeta 的语句:mvmeta(formula,S,data,subset,method=“reml”,bscov=“unstr”,model=TRUE,contrasts=NULL,offset,na.action,control=list())
其中formula 表示结局变量名称(即YAC、YBC);S 表示研究内(协)方差(即SAA、SAB、SBB);data 表示数据集名称;method 表示所用的估计方法:固定效应模型时选择FIXED;随机效应模型时则选择
限制性最大似然估计(REML)、最大似然估计(ML)、矩估计(MM)、方差成分法(VC)的其中之一,默认为REML。由输出结果中Q 检验的P 值和I2 统计量来判断异质性以及选择何种效应模型。
mvmeta包中主要提供了多变量Meta分析与多变量的Meta 回归,另外也提供了单变量的Meta 分析和Meta 回归。但对于后两者,在R 语言中的metafor、meta、rmeta 及metalik 等包提供了更多、更详尽和有效的功能。多变量Meta 程序为library(mvmeta),调用mvmeta软件包。
model<-mvmeta(cbind(Ya,Yb),S=S,data=cirrhosis)
model <- mvmeta(cbind(Ya,Yb)~X,S=S,data=cirrhosis),此处X代表协变量。
model<-mvmeta(Y,S=S,data=cirrhosis),此处Y为单变量的效应量,S为效应量方差。
model<-mvmeta(Y~X,S=S,data=cirrhosis),此处X代表协变量。
运行以上程序后,最后将结果输出。
单变量和多变量Meta分析都是采用ln(OR)值做分析。单变量Meta分析时YAC和YBC的Q检验P 值均小于0.05,I2统计量分别为57.7%和77.8%。多变量Meta分析Q检验P<0.05,I2统计量为73.9%。可知两种Meta 分析均存在异质性,都用随机效应模型。估计方法选择默认的REML法。
表2 是单变量Meta 分析结果,可得:AC 与BC的OR 值及95%可信区间分别为0.5281(0.2802,0.9955)、0.5406(0.3095,0.9443),表明初次出血的危险性由于干预而降低,即β⁃受体阻滞剂、硬化疗法可以预防肝硬化出血,两者为保护因素。
多变量Meta 分析的结果:YAC 为-0.6755(-1.3073,-0.0438),YBC 为-0.5938(-1.1444,-0.043 2),研究间相关系数为0.436 5(见表3),A组与B组的治疗效果呈正相关。OR 值及95%可信区间分别为0.508 9(0.2705,0.9571)、0.5522(0.318 4,0.957 7),多变量Meta 分析的结果说明β⁃受体阻滞剂预防肝硬化出血的效果是最好,其次是硬化疗法。OR 值的95%可信区间不包含1,上下限均小于1,说明两种疗法与对照组比较的初次出血危险性均小于1,差异有统计学意义。
最后,如果屏幕前的你对R语言学习还有什么问题或者看法,可以在我的公众号(全哥的学习生涯)给我留言,公众号里也有我的个人联系方式,我也希望可以结合更多志同道合的伙伴。
感谢你的阅读。
比如 Horticulture Research 中的论文 Comparative analysis of long noncoding RNAs in angiosperms and characterization of long noncoding RNAs in response to heat stress in Chinese cabbage 方法部分写道
这里相当于是计算两个数据集中的变量之间的相关性,之前发现 correlation 这个R包里的函数 correlation() 可以做
但是这里遇到了一个问题
关掉这个报错界面以后就会提示
暂时还不知道如何解决,自己搜索了一下暂时还没有找到解决办法
只能把输入法切换成中文,然后一次性把函数名输入完
计算相关系数和P值
结果如下
但是mRNA的表达量有上万个,用这个函数计算的时候是非常慢的
找到了另外一个函数是 Hmisc 这个包中的 rcorr() 函数
这个速度快很多,但是他不能计算两个数据集之间变量的相关性,
这样的话可以先计算,然后再筛选
这个函数要求的输入数据是矩阵格式
自定义函数将这个结果转换成一个四列的数据框格式
最后用变量名去匹配
两个矩阵之间的相关性热图这么容易画的吗?零基础学习R语言之相关性分析2_哔哩哔哩_bilibili
psych 这个包里的 corr.test() 函数也是可以直接计算两个数据集变量之间的相关性的,这个结果了也有显著性检验的p值
但是这个如果数量量比较大的话速度也很慢
小明的数据分析笔记本