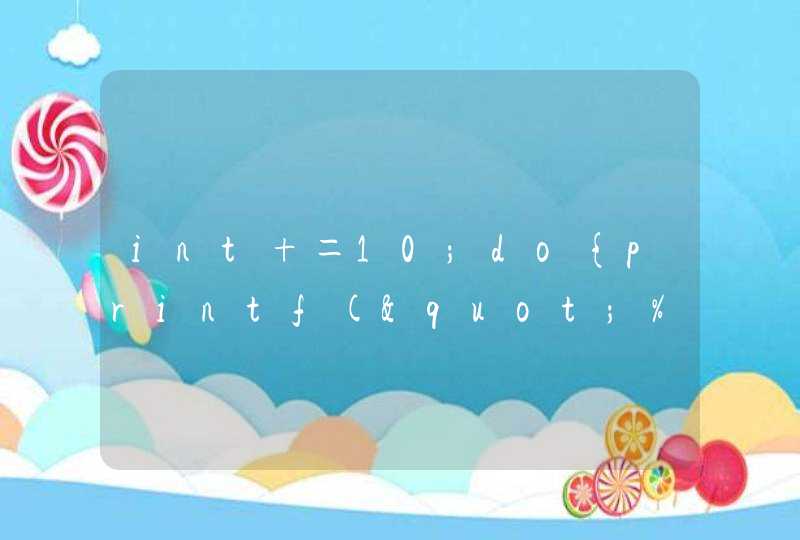VIF分析过程中,需要基于RDA/CCA进行相关性分析,RDA/CCA 模型选择原则同RDA/CCA分析。
RDA分析即冗余分析,是环境因子约束化的PCA分析,可以将样本和环境因子反映在同一个二维排序图上,从图中可以直观地看出样本分布和环境因子间的关系。CCA分析是基于对应分析发展而来的一种排序方法,将对应分析与多元回归分析相结合,每一步计算均与环境因子进行回归,又称多元直接梯度分析。此分析主要用来反映菌群与环境因子之间关系。RDA是基于线性模型,CCA是基于单峰模型。分析可以检测环境因子、样本、菌群三者间的关系或者两两之间的关系。
RDA分析是一种约束性对应分析方法,常采用欧氏距离(Euclidean distances)进行分析。但是欧氏距离并不适用于一些数据类型,采用db-RDA分析可以解决数据类型的限制,并用于分析物种与环境因子之间的关系。
db-RDA(distance-based redundancy analysis)是一个五步分析过程:
Mantel test是检验两个矩阵相关关系的非参数统计方法。Mantel test多用在生态学上检验群落距离矩阵(比如UniFrac distance matrix)和环境变量距离矩阵(比如pH,温度或者地理位置的差异矩阵)之间的相关性(Spearman 等级相关系数等)。Partial Mantel test在控制矩阵C的效应下,来检验A矩阵的残留变异是否和B矩阵相关。该分析输入两个数值型矩阵,第三个控制矩阵可通过选择因子来确定。
软件:Qiime
相关性 Heatmap分析通过计算环境因子与所选物种之间的相关性系数(Spearman等级相关系数、Pearson相关系数等),将获得的数值矩阵通过Heatmap图直观展示。通过颜色变化反映二维矩阵或表格中的数据信息,颜色深浅表示数据值的大小,它可以直观地将数据值的大小以定义的颜色深浅表示出来。
软件:R(pheatmap package)。
线性回归(Linear Regression)是利用数理统计中回归分析,来确定一个或多个自变量和因变量之间关系的一种统计分析方法。环境因子排序回归分析,常根据Alpha多样性或Beta多样性分析结果,以各样本对应的Alpha多样性指数,或在Beta多样性分析结果PC1轴上的分值为y轴,以该样品对应的环境因子(如pH、温度等)为x轴做散点图,并进行线性回归(Linear Regression),标注R2,可用于评价二者间的关系。其中R2为决定系数,代表变异被回归直线解释的比例。
VPA(Variance partitioning analysis)方差分解分析,可用于定量评估两组或多组(2~4组)环境因子变量对响应变量(如微生物群落差异)的单独解释度和共同解释度,常配合RDA/CCA使用。
分析软件:R语言vegan包中vpa分析。
MaAslin(Multivariate Association with Linear Models)分析是一种通过线性模型挖掘环境因子(如临床数据标等)与微生物群落物种或功能相对丰度(data)间相关性的分析方法,结果为一个环境因子对应一个物种或功能相对丰度的线性关系,而与其他环境因子无关。环境因子可以是连续型数据(例如年龄和体重),布尔型数据(性别),或离散型/因子数据(队列分组和表型),data代表的物种相对丰度或功能相对丰度百分比,其分布一般不符合正态分布,因此在该分析过程中data要进行反正弦平方根标准化,而环境因子通过boosting算法得到潜在的与data有联系的环境因子,在构成多元线性模型之前,还需要对环境因子和data进行质检,剔除一些异常值,和一些低丰度或无差异的值。最后以环境因子为预测值,data为响应量构建多元线性模型,计算对应的相关系数,进行相关性显著检验。相关系数大于0时,表示正相关;小于0,表示负相关,当对应的显著性检验值p值和q值符合阈值时,非连续型数据绘制箱式图,连续型数据绘制其中线性拟合度最高的散点图。
Procrustes分析(Procrustes analysis)是一种用来分析形状分布的方法。数学上来讲,就是不断迭代,寻找标准形状(canonical shape),并利用最小二乘法寻找每个样本形状到这个标准形状的仿射变化方式。普氏分析可基于不同多元数据集的排序构型(≥2组),通过平移、旋转、缩放等转换方式,实现最大叠合(maximal superimposition),用于不同数据集的对比分析。排序方法可选择PCA、PCoA等。
为了展示相同个体不同组学数据,或者不同表型数据之间的相似性关系以及之间的差异性,所以采用了普氏分析。这里展示的是如何将两组数据进行普氏分析并进行可视化
1 | 微生物种水平丰度表
2 | 食物数据的非加权unifrac矩阵
1 | 首先以食物和微生物为例
2 | 营养与微生物之间的普氏分析
3 | 谷物类数据和微生物数据(欧几里得距离)
4 | 水果数据和微生物数据
5 | 出图,大功告成
[1] https://www.cell.com/cell-host-microbe/fulltext/S1931-3128(19)30250-1