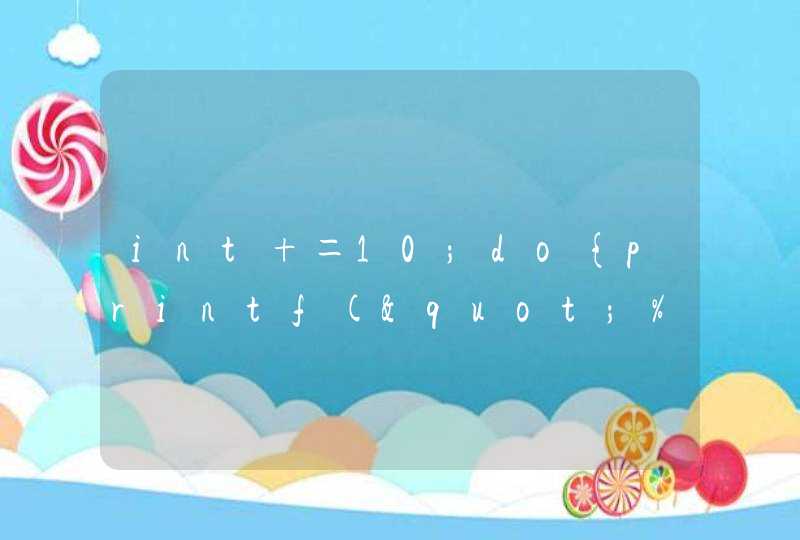%>%是管道符的意思,把左边的输出(不包括 <- 之前的)当成右边的输入。
都可以shift + alt + 上下 :快速复制粘贴
alt + 上下 :移动行
ctrl + alt + 上下 :多重光标
首先选中要注释掉的行,然后按Ctrl+shift+C ,这样就注释掉了。
sessionInfo()
.libPaths()
一篇关于包的博客
library(installr)
updateR()
COS中文论坛 统计之都旗下的论坛网站(d.cosx.org),它和其主站(cosx.org)一 起,是一个致力于推广与应用统计学知识的网站和社区。
1 help("t.test")
2 ?t.test
3 help.search("t.test")
4 apropos("t.test")
5 RGui>Help>Html help
6 查看R包pdf手册
getwd() 显示工作目录
setwd() 设定工作目录
list.files() 列出目录或文件夹下的文件
demo( ) 显示R的基本程序包
example( ) 显示在线帮助的例子
example(barplot)
可以把若干行命令保存在一个文本文件(比如Eg3.R)中,然 后用source函数来运行整个文件: source("E:/R demo/Chapter1-Eg3.R")
sum, mean, var, sd, min, max, range, median, IQR(四分位间距)等为统计量, sort,order,rank与排序有关, 其它还有ave,fivenum,mad,quantile, stem等
-1:1/0 当中/是优先级靠后的操作。相当于c(-1,0,1)/0
names(df) <- c("male", "female", "unknown")
对于矩阵,我们可以使用属性rownames和colnames来访问行名和列名。
我们也可以先定义矩阵x然后再为dimnames(x)赋值:
数值型数据 :1.2345e30
复数常量就用3.5-2.1i
缺失值:NA(Not Available)
是否含有缺失值:
NaN表示不确定的数
• NaN属于NA的一种
• NA不是NaN
注意下面例子的比较 :
assign("x1", c(1, 2))
sort(x)返回x的元素从小到大排序的结果向量。
x=c(2,10,6,8,4,5)sort(x) [1] 2 4 5 6 8 10 order(x)返回使得x从小到大排列的元素下标向量(x[order(x)]等效于sort(x))。
此外numeric(n)可以产生一个长度为n的零向量(numeric(n)是一个 很好用的外部存储器)
paste函数用来把它的自变量连成一个字符串,中间用空格分开
Re( )计算实部,Im( )计算虚部, Mod( ) 计算复数模,Arg( )计算复数幅角。
v为一个向量,取值在-length(x)到-1之间,表示扣除相应 位置的元素。例如:
可以用x[]的写法:
R的对象有两个基本的属性:类型属性(mode)和长度属性(length)。
长度为零的向量 numeric( ) 或者 numeric(0) character( ) 或者 character(0)
数组(array): 带多个下标的类型相同的元素的集合,
函数matrix():用于构造二维数组,即矩阵
函数factor( )用来把一个向量编码成为一个因子。
可以自行指定各离散取值水平(levels),不指定时由x的不同值来求得。
• labels可以用来指定各水平的标签,不指定时用各离散取值的对应字符串。
• exclude参数用来指定要转换为缺失值(NA)的元素值集合。
• ordered取真值时表示因子水平(Levels)是有次序的
因子可以用来作为另外的同长度变量的分类变量,使用tapply() 函数可以完成分类统计
nchar()这个函数简单,统计向量中每个元素的字符个数
tolower()和toupper()可以进行大小写字母的转换
chartr()把字符串里的元素,按要求进行转换
拆分字符串用strsplit()函数,strsplit得到的结果是列表,后面的处理要调用列表
其任何一个语句都可以看成是一个表达式。
表达式之间以分号分隔或用换行分隔。
表达式可以续行,只要前一行不是完整表达式,则下一行为上一行的继续。
线性回归模型:
lm()函数的返回值叫做模型拟合结果对象,本质上是一个列表, 有model 、coefficients、residuals等成员。lm()的结果显示十分 简单,为了获得更多的拟合信息,可以使用对lm类对象有特 殊操作的通用函数,这些函数包括:
add1 coef effects kappa predict residuals alias deviance family labels print summary anova drop1 formula plot proj
加号+或 者减号-,表示在模型中加入一项或去掉一项,第一项前面如果是加号可以 省略
在非交互运行(程序)中应使用print()来输出。
• digits参数指定每个数输出的有效数字位数;
• quote 参数指定字符串输出时是否带两边的撇号;
• print.gap参数指定矩阵或数组输出时列之间的间距
也用来输出,但它可以把多个参数连接起来再输出(具有paste() 的功能)。例如:
读取文件:
strsplit()得到的结果是 列表。
grep() 和 grepl()
sub()和gsub()
但严格地说R语言 没有字符串替换的函数,因为R语言不管什么操作对参数都是传值不传址,区别如下:
用substr()和substring() 可以通过位置进行字符串拆分或提取,两者的参数设置基本相同:
strtrim() 函数可以用于将字符串修剪到特定的显示宽度通过位置进 行字符串拆分或提取:
由于日期内部是用double存储的天数,所以是可以相减的。
weekdays ( )取日期对象所处的周几;
months ( )取日期对象的月份;
quarters ( )取日期对象的季度;
其任何一个语句都可以看成是一个表达式。
表达式之间以分号分隔或用换行分隔。
表达式可以续行,只要前一行不是完整表达式,则下一行为上一行的继续。
quantile(x, probs=seq(0,1,0.25), na.rm=FALSE, names=TRUE, type=7, …)
probs给出相应的百分位数,默认值是0,0.25,0.5,0.75,1;na.rm是处 理缺失数据的,na.rm=TRUE时,NA和NaN将从数据中移走,向量取值中 若有NA或NaN,要添加这一参数,否则会出错;names若为TRUE,返回 值当中有names这个属性"type是取值1-9的整数,选择了九种分位数算法 (具体算法见帮助文件)中的一种。
数据的分布主要考察分布函数(p), 密度函数(d), 分位数函数(q)及产生随机数(r)
以正态分布为例:
hist(x, breaks="Sturges", freq=NULL, probability=!freq,… )
break规定了直方图的组距(必须覆盖数据的范围);freq是逻辑变量,TRUE是频率直方图, FALSE是密度直方图;probability和freq相反,TRUE是密度直方图,FALSE是频率直方图
其形式为 coplot(y ~ x | z),其中x 和y是数值型向量,z是同长度的因子。 对z的每一水平,绘制相应组的x和y的散点图
R缺省的图形边空常常太大,以至于有时图形窗口较小时边空占了整个图形的很大一部分。
R可以在同一页面开若干个按行、列排列的窗格,在每个窗格中可以作一 幅图。每个图有自己的边空,而所有图的外面可以包一个“外边空”。
一页多图用 mfrow 参数或 mfcol 参数规定,如
函数 mtext 用来在外边空加文字标注。其用法为
在多图环境中还可以用 mfg 参数来直接跳到某一个窗格,比如
可以不使用多图环境而直接在页面中的任意位置产生一个窗格来绘图,参数为 fig ,如:
先用as.factor()转化成因子。因为levels()函数里面必须是因子。
dat$Genre没有转化成因子形式,as.factor(dat$Genre)就可以了
该消息表明文件的最后一行不以行尾 (EOL) 字符结尾(换行符 ( \n ) 或回车 + 换行符 ( \r\n ))。此消息的初衷是警告您该文件可能不完整;大多数数据文件都有一个 EOL 字符作为文件中的最后一个字符。
这是因为R读取文件的时候,是一整段character,所以它只会返回1,适当给他分一下段。
#PCoA 分析在R语言中进行主要依赖于以下得包,进行这个分析得主要可以应用于形态学数据得相似与差异性分析。library(ade4)
library(ggplot2)
library(RColorBrewer)
library(vegan)
这里我们使用R自带得数据iris
data(iris)
在R语言中通常都会使用这个数据进行案例分析
#iris
data(iris)
iris
data01<-iris[,-5]#数据预处理,去掉最后一列得数据标签
data01
dis01<-vegdist(data01,method = "euclidean")#这里是为了算矩阵距离,方法根据数据选择合适得方法
dis01
pcoa1<- dudi.pco(dis01, scan = FALSE,nf=3)#进行PCoA分析
pcoa1
pcoa1_eig<-pcoa1$eig[1:2]/sum(pcoa1$eig)#算一下前两列对整个数据得解释比例
pcoa1_eig
samplesite1<-data.frame({pcoa1$li})[1:2]#将前两列的数据分析结果放到sample_site1里面
sample_site1
sample_site1$names<-rownames(sample_site1)#设置名称
sample_site1$names
iris$Species
sample_site1$level<-factor(iris$Species,levels = c("setosa","versicolor","virginica"))#设置level的标签
sample_site1$level
names(sample_site1)[1:2]<-c("PCoA1","PCoA2")
p<-ggplot(sample_site1, mapping=aes(PCoA1, PCoA2,color=level))+theme_classic()
p<-p+geom_point()#绘制散点图
p
下图显示内置数据集colon,病人rx处理分为三组(下图第三列),对照组: Obs ,处理组一: Levamisole (Lev) ,处理组二: Levamisole + 5-fluorouracil (Lev+5FU)# loads dplyr
library(dplyr)
# core survival analysisfunctions
library(survival)
# recommended forvisualizing survival curves
library(survminer)
#加载内置colon数据集
data(colon)
#list directory contents
ls(colon)
得到如下图:
#创建生存对象
fit <- survfit(Surv(time, status)~rx, data=colon)
#level()是为了看分组水平情况,以确定对照组,一般level()之后第一个为对照组
levels(colon$rx)
ggsurvplot(fit,risk.table=TRUE,#生存统计统计表
conf.int=TRUE,#添加置信区间带
palette = c("skyblue","green","red"),#颜色设置
pval=TRUE,#log-rank检验
pval.method=TRUE)#添加检验text
至于是treatment中的哪一组与Obs相比,显著性,差异性更大,需要查看 Lev 和 obs 对比的p值及HR,以及 (Lev+5FU) 和 Obs 对比的p值及HR,评价分组的治疗效果
#Cox Regression,评价rx分组后治疗效果
fit1<-coxph(Surv(time, status)~rx, data=colon)
fit1