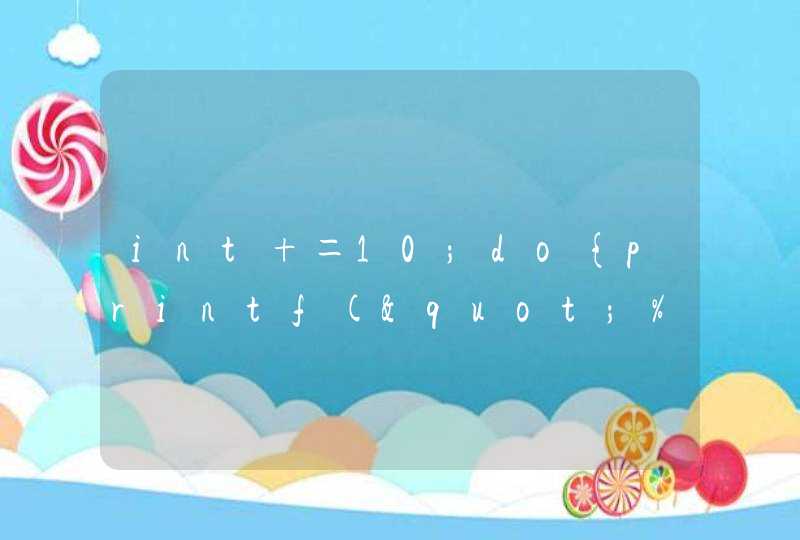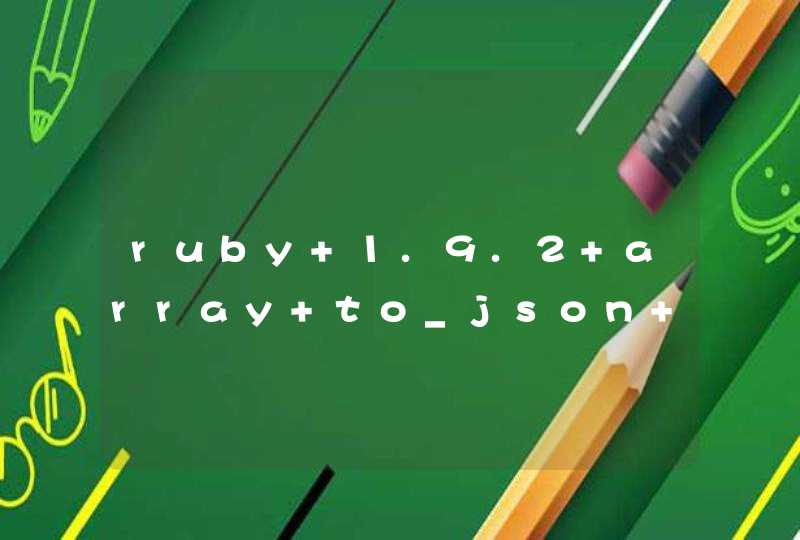安装R包报错的问题从一开始学生信就一直存在着,但是没有专门整理一下,前两天安装 CHIPseeker 的时候实在受不了了,因为碰见了好多坑,于是在这里专门整理一下,方便自己和他人查看
到这里已经没太有耐心了,然后开始查原因,后面应该加一个 type = "binary"
再试一下
成功解决,安装 XML 时同样也是这个问题 ,跟上面是一种解决方式
安装 TxDb.Hsapiens.UCSC.hg19.knownGene 就不能用 biocLite 了
解决办法
解决:去 lib_PATH下把lock文件删掉
这里是 问题 5 的 参考
老版本R,在这里MARK以下
虽然讨论了一些R包安装问题,但这应(jue)该(dui)不会是我最后一次安装报错,以后再遇到会更新的
将R语言安装包拖入虚拟机中Ubuntu Kylin 16.04系统中:
然后查找语言包的位置:
解压文件:
进入解压后的文件夹:
源码编译安装第一步:检查环境以及依赖
报错排查:
缺少编译器,解决办法:
另外还需要安装gcc等工具:
解决方案:安装readline
解决方案:安装libxt-dev
解决方案:源码编译安装zlib
解决方案:源码编译安装bzip2
解决方案:安装xz
解决方案:pcre版本过低,安装pcre-8.41.tar.gz
错误总算排查完了,继续安装R
源码编译安装第一步:检查环境以及依赖
读取表格 坑1:列名称不对,使用了R不能识别的列名1 | follow_table <- read.csv("follow_table.csv", header = T)
返回
follow_table <- read.csv("follow_table.csv", header = T)
Error in make.names(col.names, unique = TRUE) :
invalid multibyte string 2
返回表格,把列名中含数字的都去掉,改成字母+下划线就好了。
读取表格坑2 :
orignal_table <- read.csv("original_table.csv",header = T)
Error in read.table(file = file, header = header, sep = sep, quote = quote, :
列的数目比列的名字要多
后来看了,表格为逗号分隔符,加上 sep = "," ,还是没好,把表头 设置成 header = F 就好了
orignal_table <- read.csv("original_table.csv",sep=",", header = F)
这样读取的表格还是不行,因为第一列的列名为乱码。
百度搜索了解决办法,[R语言读取csv文件,第一列列名出现乱码的解决方法] ( Rhttps://blog.csdn.net/weixin_45075290/article/details/90695638 )
原来是因为只是把表格另存为csv是不行了,需要把表格导出为CSV的格式。
两个表格通过同一个列名合并,最终得到的是其同一列名交集的最终对应的变量。